自20世纪20年代人类发现胰岛素以来,蛋白多肽类药物在体液调节、抗菌、抗炎、抗病毒及抗肿瘤等临床应用方面的重要性愈加显著[1-3]。截至2017年,全世界已有60多种多肽类药物获批上市,并以平均每年1种的速度持续增长[4-5]。虽然以重组表达为代表的现代分子生物学技术能够高效生产重组蛋白且可掺入个别非天然氨基酸,但难以合成含有多种非天然氨基酸的人工设计多肽与蛋白质[6-7]。目前多肽固相合成法仍是便捷获取非天然多肽的主要途径,然而该方法能够合成的多肽长度一般限于30~50个氨基酸残基,研究人员需要将多个多肽片段顺次拼接从而获得完整目标蛋白[8-9]。除此之外,许多待研究的多肽或蛋白质分子需要在特定位点连接寡糖链、脂类分子、核酸、荧光分子或另一个蛋白质分子等多样的功能基团。为了降低这些蛋白质的合成难度与成本,研究者采取了半合成策略,即利用化学方法合成一段带有功能基团的短肽,再将该片段与重组表达的蛋白质连为一体[10-11]。两段合成多肽的拼接以及蛋白质半合成均极具挑战,其难点在于需要保证酰胺缩合反应的区域选择性,同时必须抑制末端氨基酸残基的外消旋化副反应[12]。因此,探索具有严格区域选择性且尽可能避免外消旋的多肽拼接方法成为了蛋白质化学合成与修饰领域近年来的研究焦点。
现有的多肽拼接方法分为化学法与酶促法两大类。化学法包括自然化学连接(native chemical ligation,NCL)[13]、无痕施陶丁格连接(traceless Staudinger ligation)[14]、酮酸-羟胺连接(ketoacid-hydroxylamine ligation,KAHA)[15]、丝氨酸/苏氨酸连接(serine/threonine ligation,STL)[16]以及二硒醚-硒酯连接(diselenide-selenoester ligation,DSL)[17]等(图1)。这些化学方法均采取了相似的策略,即两条多肽片段的末端化学基团发生选择性反应形成共价键,之后通过分子内重排形成肽键[12]。由Kent团队提出的NCL是目前应用最为广泛的一种方法,该方法的原理为将一条多肽的C端活化为硫酯形式,而另一条多肽的N端第一个残基固定为Cys,二者经过硫醇-硫酯交换与分子内重排两步反应形成肽键[18]。多肽硫酯可通过Boc固相合成法(保护基为叔丁氧羰基)直接合成,也可通过Fmoc固相合成法(保护基为9-芴甲氧羰基)制备多肽酰肼,再经过一步反应生成叠氮[19]或吡唑[20]中间产物,加入硫醇得到对应的多肽硫酯。若待连接片段为重组蛋白,则需要通过分子生物学方法在蛋白N端添加Cys残基,或是将蛋白质C端活化为硫酯形式。目前最常用的EPL策略需在蛋白质C端融合表达内含肽(Intein),之后在反应体系中加入苯硫酚以进攻内含肽与目标蛋白的结合处,从而形成硫酯[21]。NCL方法的主要缺陷在于Cys是天然蛋白质中丰度最低的氨基酸种类之一,这严重限制了拼接位点的选择范围,尽管连接后可通过脱硫处理将Cys变为Ala,但如果存在其他非连接位点的Cys残基,又需要增加保护与脱保护操作,这些步骤都会降低最终产物的收率[22]。此外EPL策略最初使用的内含肽长约140个氨基酸残基,虽然后续研究缩短了融合表达内含肽片段的长度[23],仍可能对重组蛋白的表达产生较大影响。
图1
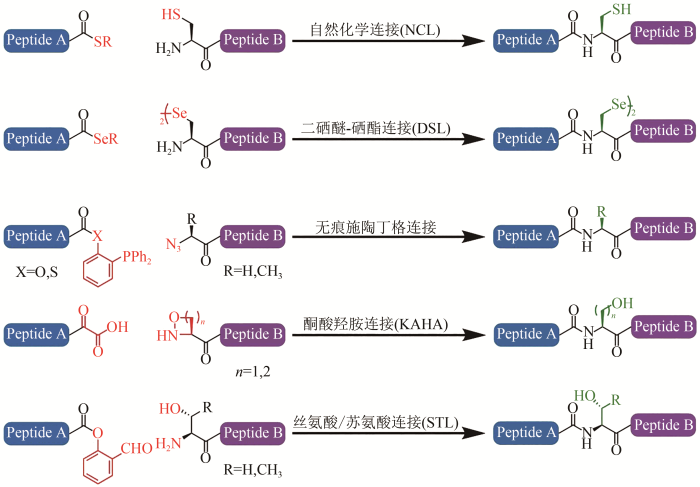
与化学法的原理不同,酶促法反应的区域选择性与立体选择性来源于酶活性中心的空间位阻以及基团间的非共价作用,满足多肽拼接在区域选择性及抑制外消旋方面的基本要求。目前研究较深的酶促多肽拼接策略主要有三种,分别使用Sortase A转肽酶、Butelase 1转肽酶以及Subtilisin人工连接酶,这些方法在拼接位点限制、蛋白质表达难度、酶活性等方面凸显出不同的优势,但在各个应用领域又分别有各自的限制,下文将系统阐述与比较。
1 多肽连接酶性质及其应用
1.1 Sortase A转肽酶
SortaseA(SrtA)是源自革兰氏阳性菌的一种转肽酶,其中来源于金黄色葡萄球菌(Staphylococcus aureus)的SrtA转肽酶应用最为广泛,该酶识别蛋白的LPXTG序列(X为任意氨基酸残基),切断苏氨酸和甘氨酸残基之间的肽键并形成酶-底物中间体,随后位于肽聚糖的寡聚甘氨酸肽桥进攻中间体,与目标蛋白C端之间形成新的肽键从而实现目标蛋白在细胞壁表面的锚定[24]。此外还存在识别LPXTA序列的SrtA转肽酶,这种来自化脓性链球菌(Streptococcus pyogenes)的转肽酶常出现在与金黄色葡萄球菌来源的SrtA转肽酶联合使用的场合,两种酶对拼接位点的序列识别具有正交性[25]。SrtA转肽酶可通过重组表达来大量制备,现已实现商品化,这让不同领域的研究者均能尝试以此为基础开发新型生物技术[26]。与内含肽介导的EPL策略相比,使用SrtA转肽酶催化蛋白质拼接时,仅需在目标蛋白末端融合表达长度仅有几个氨基酸残基的标签序列,虽然最终会留下一段“疤痕序列”[图2(a)],但与融合表达内含肽的策略相比,已大幅降低了蛋白质表达与折叠可能受到的影响,并且底物末端无需活化,获取底物的难度与成本较低。
图2
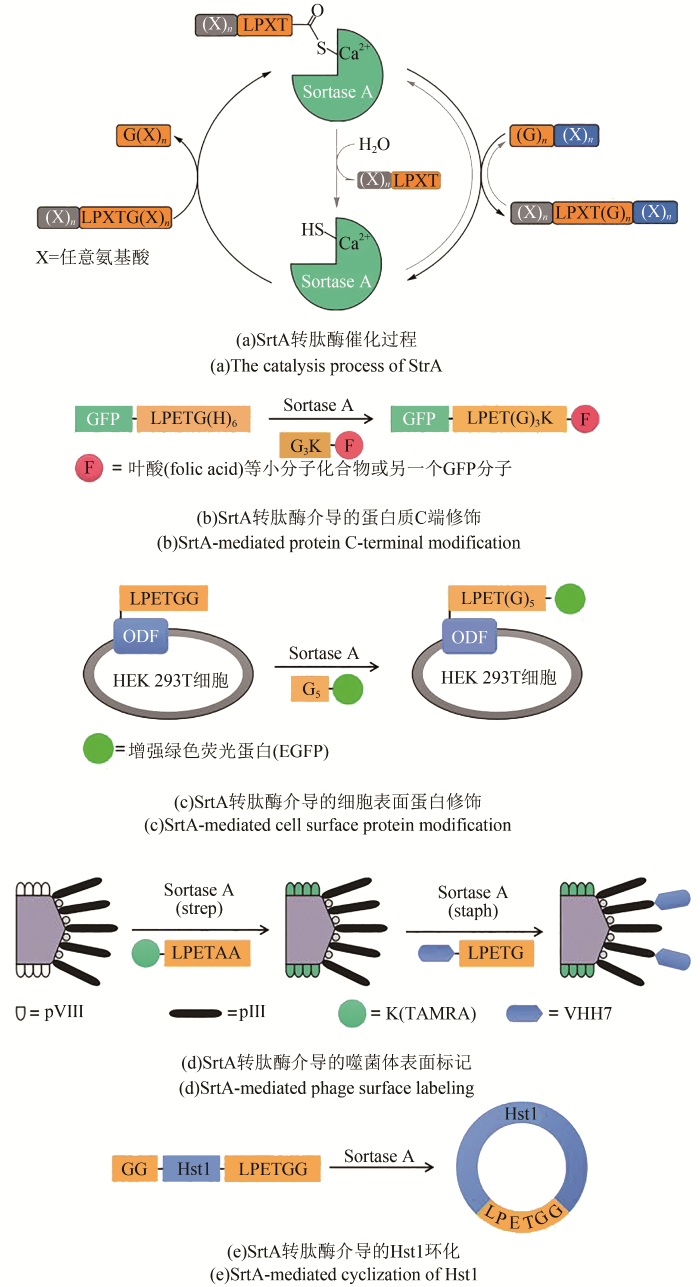
2004年,Mao[27]等首次利用SrtA转肽酶进行蛋白修饰,在绿色荧光蛋白(green fluorescent protein,GFP)的C端融合表达LPETG(H)6标签,再通过转肽反应将叶酸等小分子化合物或另一个GFP分子连接至目标蛋白C端[图2(b)],此后SrtA转肽酶蛋白拼接技术很快被应用于多个生物学研究方向。细胞表面蛋白参与了细胞生长、分化、识别等众多生物学过程,一直作为重要的药物靶点受到广泛关注,Tanaka等[28]将LPETGG序列添加至重组破骨细胞分化因子C端并运用SrtA转肽酶方法进行修饰[图2(c)],为细胞表面蛋白的时空动力学表征提供了新的研究思路,同样的方法亦可用于细胞表面蛋白的N端修饰[29]。噬菌体表面展示技术是当下建立抗原抗体库、筛选药物等研究的常用手段,Hess等[30]提出了组合应用衣壳蛋白的策略,通过在M13噬菌体衣壳蛋白pIII与pVIII的N末端分别添加寡聚Gly和寡聚Ala(G5-pIII-A2-pVIII),先利用化脓性链球菌(Streptococcus pyogenes)来源的SrtA转肽酶[Sortase A(strep)]将四甲基罗丹明(tetramethylrhodamine,TAMRA)修饰的七肽(KLPETAA)选择性连接至pVIII,再利用金黄色葡萄球菌(Staphylococcus aureus)来源的SrtA转肽酶[Sortase A(staph)]将带有五肽标签(LPETG)的骆驼科重链抗体7(camelid heavy-chain antibody 7,VHH7)选择性连接至pIII,拓宽了噬菌体表面展示技术的功能范围[图2(d)]。与蛋白修饰相比,SrtA转肽酶在环肽合成方面的成果较少,主要原因是连接后留下的“疤痕”较长,另外待环化多肽的长度一般需要超过19个氨基酸残基,否则将倾向于肽段寡聚[31]。目前SrtA转肽酶方法合成环蛋白的代表案例包括人唾液肽组蛋白Hst1[图2(e)][32]、重组胱氨酸结环肽rMCoTI-II[33]以及向日葵胰蛋白酶抑制剂SFTI-1[34]等,环化后蛋白的热稳定性以及抗蛋白酶水解能力均有不同程度的提升。
SrtA转肽酶的主要不足之处在于其催化的连接反应是可逆的,因而需要较高的多肽底物浓度以保证连接效率,此外酶的催化速率偏慢,酶用量一般为蛋白底物物质的量的0.1~1倍[10,35-36]。当下研究者的一种优化思路是将SrtA转肽酶与其他化学或酶学方法结合以取长补短,例如Muir团队提出了基于内含肽自剪切技术与SrtA转肽酶转肽反应的TAIL策略,该策略可用于细胞核等复杂生物环境下的蛋白质末端不可逆连接,“疤痕”序列仅含一个Cys残基[37]。清华大学刘磊团队[38]则用肼或肼衍生物替代寡聚甘氨酸底物进攻SrtA转肽酶-底物中间体,产物变为不再被SrtA转肽酶识别的蛋白质酰肼,由此可制备NCL方法所需的蛋白质硫酯,或是在蛋白质末端引入炔基、叠氮等功能基团,通过点击化学反应(click reaction)完成二次修饰。SrtA转肽酶的技术扩展案例为蛋白质合成与修饰领域指出了一个潜在的发展方向,即搭配使用现有的化学与酶促方法,建立充分发挥多种方法优势的多肽连接策略。
1.2 Butelase 1转肽酶
Butelase 1转肽酶是热带药用植物蝶豆(Clitoria ternatea)合成环肽的过程中催化多肽环化的连接酶,识别多肽C端的N/D-HV序列并切断N/D的C端肽键形成酶-底物中间体,之后底物N端进攻中间体从而完成多肽的环化[39-40]。Butelase 1转肽酶的连接位点最终仅留下一个氨基酸残基(Asn或Asp)的“疤痕”[图3(a)],底物N端的序列限制也比SrtA转肽酶低得多,因此Butelase 1转肽酶在环肽合成方面更具优势[40-41]。Tam团队[42]利用Butelase 1转肽酶合成了多种环蛋白,例如环状细菌素AS-48[图3(b)]、Uberolysin和Garvicin ML,其中AS-48对包括李斯特菌(Listeria monocytogenes)在内的多种致病细菌具有优秀的抑制效果,其最小抑菌浓度低至0.1 μmol/L数量级,为开发针对“超级细菌”的特效药物指明了新方向。可被Butelase 1转肽酶环化的多肽长短不一,大到含有约250个氨基酸残基的GFP(环化速率约为SrtA转肽酶的20 000倍),小到九肽,更短的多肽片段则一般会先寡聚后成环[43]。
图3
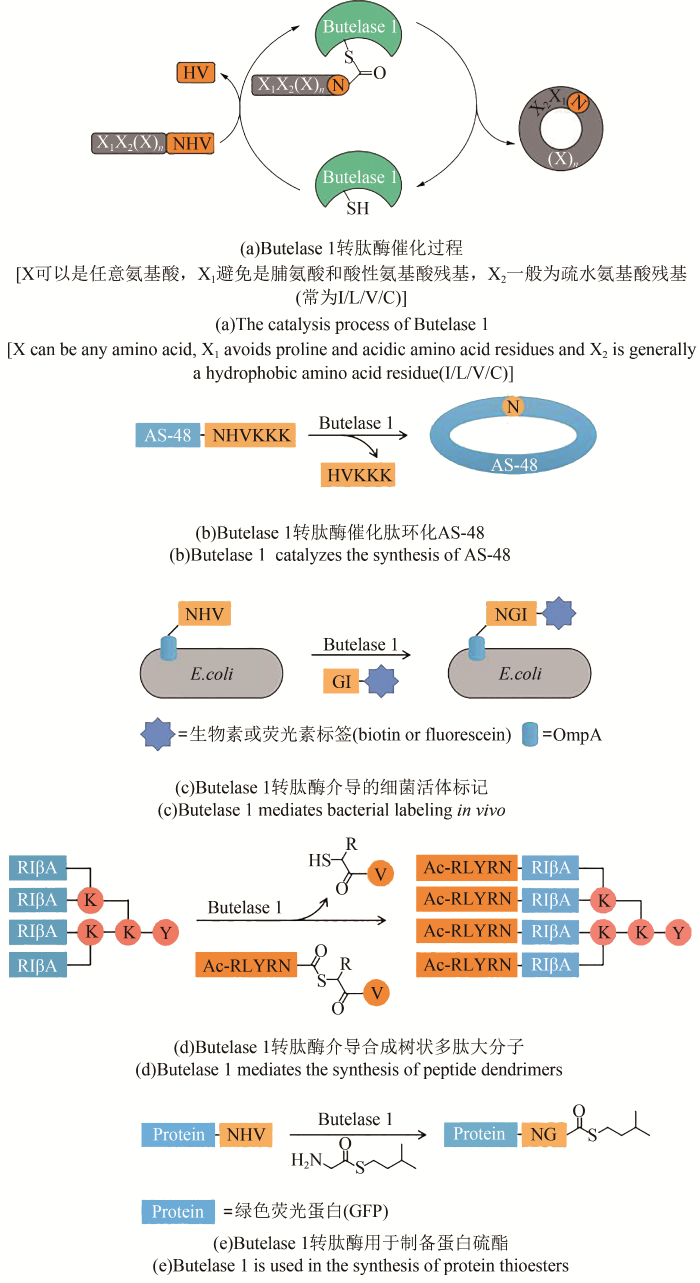
实验表明,Butelase 1转肽酶催化的多肽首尾环化反应不可逆,但两条多肽间的拼接反应是可逆的,释放的HV二肽同样可以作为亲核试剂进攻酶-底物中间体,这导致Butelase 1转肽酶和SrtA转肽酶类似,需要较高的底物浓度来保证多肽拼接效率[44]。为了解决这一问题,Tam团队[44]合成了C端序列为N-(thioglycolic)-V的多肽底物,此序列依旧能被Butelase 1转肽酶识别,但释放的硫代二肽无法进攻酶-底物中间体,改进后的方法用于泛素N端修饰时取得了高达95%的连接率[44]。不过在对连接率要求不是特别高的情况下,普通的底物已经能满足大部分需求,例如在大肠杆菌表面的OmpA蛋白C端添加NHV序列,利用Butelase 1转肽酶即可将带有甘-异亮(GI)二肽的分子探针连接至OmpA蛋白上,从而实现细菌活体标记[图3(c)][45]。当被修饰的蛋白质分子是一个以Lys为节点的多肽分支骨架时,就可以在这个“树干”上连接“枝叶”,将八条具有抗菌作用的四肽(RLYR)连接至这种骨架上[图3(d)],形成的树状多肽大分子的广谱抑菌活性相比单体有大幅提升,最小抑菌浓度降低了2个数量级[46]。此外Butelase 1转肽酶亦可用于蛋白硫酯的制备,目标蛋白C端仅需额外表达NHV三个氨基酸残基,再通过转肽反应连接多肽硫酯,无需内含肽的参与[图3(e)][47]。
1.3 Subtilisin人工连接酶
Subtilisin是来自解淀粉芽孢杆菌(Bacillus amyloliquefaciens)的丝氨酸蛋白酶,具有六个广谱的氨基酸残基识别口袋,最初作为一种具有广泛切割位点的蛋白水解酶而受到关注。早在20世纪60年代,Bender团队[51]即使用化学方法将Subtilisin的关键活性基团从Ser转变为Cys(S221C),得到的人工连接酶Thiolsubtilisin在50% DMF溶液中展现出了多肽连接酶的活性,但在水溶液中效率极为低下[52]。随着定点突变技术的发展,Wells团队[53]获得了重组表达的多肽连接酶Subtiligase,在S221C之外还引入了P225A突变来降低活性中心的空间位阻,将该酶的连接活性提高了1个数量级且水解活性降低了2个数量级,从此奠定了Subtilisin人工连接酶的基本形态。之后Wells团队[54]开展了一系列酶工程研究,先是额外引入5个突变位点(M50F/N76D/N109S/K213R/N218S)以提高连接酶的稳定性,使得连接酶在4 mol/L盐酸胍的环境中依旧能保留50%的活性;后续又在噬菌体表面展示技术[55]以及蛋白质组技术[56]的辅助下,改造得到一系列具有不同序列识别偏好的Subtiligase突变体,从而扩展P1'与P2'口袋的底物谱。
相比SrtA转肽酶和Butelase 1转肽酶,Subtilisin人工连接酶对连接位点的序列限制更少,不会出现“疤痕”序列[图4(a)],因此具有更广阔的应用范围。早在1994年,Wells团队[57]便利用Subtiligase将6个多肽片段(每段长度在11~31个残基)拼接为含有124个氨基酸残基的核糖核酸酶A,酰基供体多肽N端被异烟碱(isonicotinyl,iNOC)基团保护以防止重复拼接,每轮连接反应后去除保护基来进行下一轮的连接[图4(b)],这比NCL方法更早地实现了将多个多肽片段拼接为完整蛋白质[57]。Subtiligase还被用于环化长度在12~31个氨基酸残基的多肽[图4(c)][58],在内含肽的辅助下可以实现蛋白质C端的无痕拼接,例如Cole等[59]利用Subtiligase催化重组表达的泛素硫酯与合成的十肽-生物素(GLSGRGKGGK-Biotin)的连接,解除了酰基受体肽N端必须为半胱氨酸残基的限制[图4(d)]。不过Subtiligase目前最主要的应用方向仍是蛋白质N端修饰,Wells团队[60]建立了一个细胞凋亡相关蛋白水解酶研究平台,首先用Subtiligase将带有生物素标签的多肽连接在混合蛋白样品的N端[图4(e)],再用待研究的蛋白水解酶处理样品,酶切后用亲和素富集带有生物素标签的样品N端片段,最后进行HPLC-MS/MS分析[60]。生物素标记与亲和素富集的操作实现了酶解多肽片段的正向筛选,该平台可以帮助研究人员发现特定蛋白水解酶在细胞中的潜在靶标蛋白与切割位点。
图4
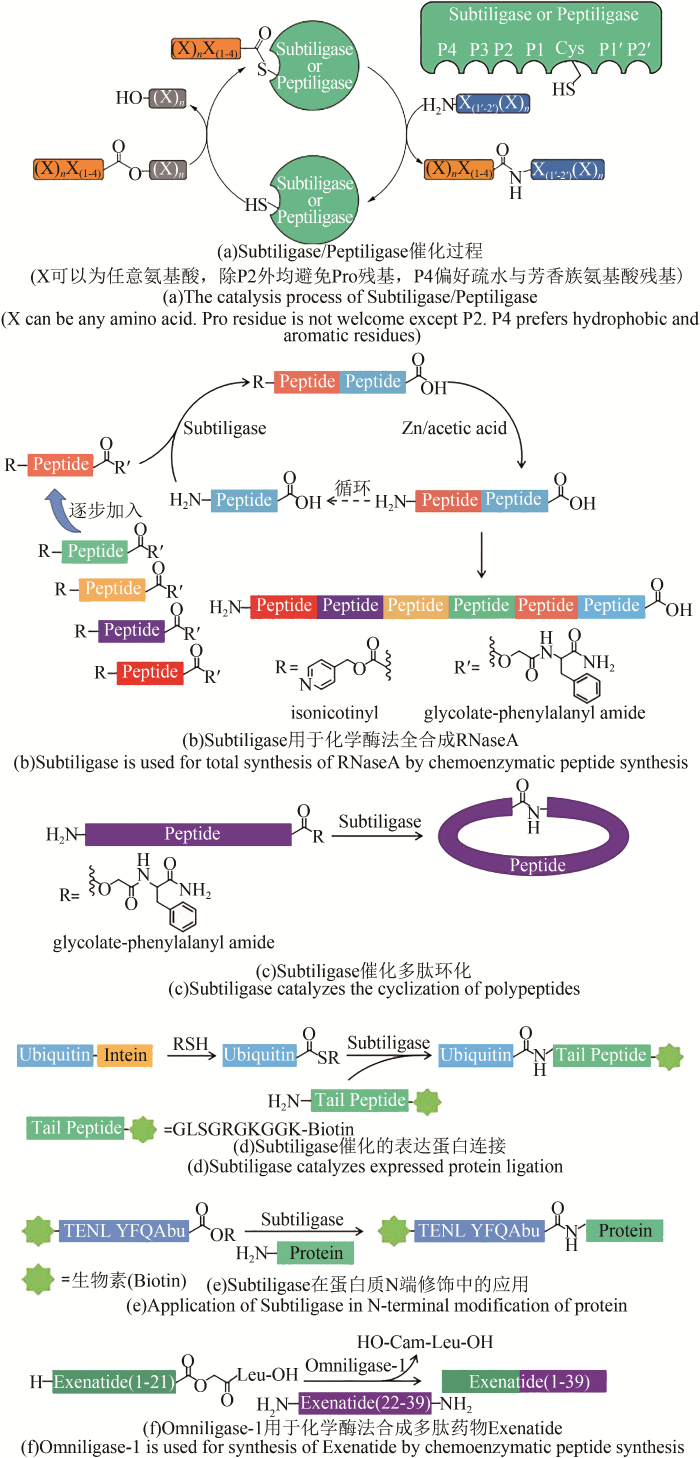
除Subtiligase之外,该家族的另一成员的研究也在近年获得突破。荷兰帝斯曼集团与格罗宁根大学合作,筛选了来自八十余种古菌和细菌的上百种Subtilisin家族蛋白酶,确定了一个不依赖钙离子且高度稳定的模板蛋白,并对其活性中心进行改造设计,创制了一种新型多肽连接酶Peptiligase[4,61]。与Subtiligase相比,Peptiligase的折叠不依赖前体肽,表达量大幅提高且连接活性更高(酶用量可低至多肽底物物质的量的万分之三),还具有适用于工业生产的极高稳定性,足以耐受60 ℃的高温、高浓度DMF与盐酸胍。后续帝斯曼集团将该部分业务独立,组建了全世界第一家应用生物法合成多肽与蛋白质的商业公司——Enzypep公司。通过对Peptiligase的深入研究与改造,Enzypep公司推出了新一代广谱连接酶Omniligase-1,为低序列限制的通用多肽拼接策略提供了优秀的工具酶[62]。在多肽药物Exenatide百克级合成过程中[图4(f)],Omniligase-1催化下的两段多肽拼接效率达到88%,总产率是原先固相合成法单次合成完整多肽的两倍,展现了该酶极为突出的工业应用价值[63]。Omniligase-1亦可与二硫键构筑[64]、点击化学[65]、肟连接(oxime ligation)[66]等方法联用以合成具有多环结构的环肽。不只是广谱连接酶,Enzypep公司还推出了针对特定药物多肽序列的连接酶,例如专门用于生产Thymosin-α1的Thymoligase,该酶在P1位特异识别带正电氨基酸,在P1'位特异识别带负电氨基酸,两段多肽底物的拼接效率高达94%,最终产率相比原有生产工艺提升了两倍[67]。Peptiligase系列连接酶在连接/水解比、反应活性、底物序列限制等多个方面突破了工业化瓶颈,使多种药物多肽的生产成本下降了60%~80%,显著的工业应用优势让Peptiligase系统被视为目前酶促多肽拼接领域最受瞩目的技术平台[68],针对不同应用方向来改造获得满足不同序列需求的突变体依旧是其未来的主要发展方向。
2 总结与展望
蛋白质重组表达与多肽固相合成技术在掺入非天然氨基酸、蛋白质表达系统适用性与多肽合成长度等方面都存在一定的限制,多肽连接技术可以将多个短肽连接成较长的肽段,同时解决了非天然氨基酸修饰与蛋白质合成长度的问题,而其中酶促多肽连接法由于其独特的区域选择性与立体选择性在蛋白质合成领域发挥着愈加重要的作用。近年来三种酶促多肽拼接策略经过优化与发展,各自具有其适合的应用场合(表1)。SrtA转肽酶可通过重组表达获得,也可以通过商业渠道购买,在蛋白质末端修饰方面便于使用,是目前应用案例最多的多肽连接酶,但在连接产物中会保留数个氨基酸残基的连接“疤痕”,不适合用于特定序列多肽或蛋白质的合成。Butelase 1转肽酶活性较高,拼接位点的序列限制比SrtA转肽酶少,连接后残留的“疤痕”较短,适合催化多肽与蛋白质的环化。然而该酶目前还无法通过重组表达制备,研究所用的酶需要从植物中获得,每千克的植物材料约能提取出5 mg酶,难以实现大规模生产,这成为该酶应用的主要限制因素。Subtilisin人工连接酶则在拼接位点序列限制方面优于前两种酶,只需要底物C末端活化为氧酯或硫酯而无需具备特定的氨基酸序列,虽然该酶的6个氨基酸残基识别口袋具有不同的氨基酸偏好性,但经过多年的研究优化,该系列酶已经产生了较多广谱突变体,并能整合具有互补选择性的连接酶突变体,建立的酶工具箱用于不同领域,功能最为全面[61]。并且Peptiligase系列商品酶是当下唯一能在工业级合成中使用的酶,不过这些商品酶大多价格高昂且序列保密,尚未普遍推广。三类酶促多肽拼接策略依旧具有较大的优化空间,研究者不仅可以联用化学和酶促方法来实现优势互补,建立不同的生物合成技术路线,还可以通过酶工程改造来提升连接酶的性能,过去三十年在这一方向已出现大量的理性设计与定向进化成果。而随着运算能力大幅提升以及先进算法不断涌现,近年来蛋白质计算设计得到了极大的发展,蛋白质从头设计的时代已经到来[69]。在蛋白质人工合成与蛋白质计算设计这两个领域相逢之际,计算机辅助手段既可以指导新型连接酶的设计与改造,提升连接/水解比,又能够帮助设计具有特殊修饰的非天然蛋白质[70],为酶促多肽拼接策略开拓崭新的应用场景。无论是化学方法与酶促策略的结合,还是人工合成与计算设计的相遇,均是不同领域的碰撞而闪现出的火花,深刻体现了学科交叉的意义与价值。相信在越来越多潜在相关领域的研究人员参与后,未来酶促多肽拼接策略的技术方法能再上一层台阶,应用于更多的多肽环化、蛋白质修饰乃至蛋白质全合成研究项目。
表1 三类酶促连接策略以及NCL方法的比较
Tab. 1
| 项目 | SrtA转肽酶 | Butelase 1转肽酶 | Subtilisin人工连接酶 | NCL | |
|---|---|---|---|---|---|
| 最初来源物种 | 金黄色葡萄球菌Staphylococcus aureus |
蝶豆 Clitoria ternatea |
解淀粉芽孢杆菌 Bacillus amyloliquefaciens |
—— | |
| 酶的获取与产量[26] |
SrtA转肽酶可通过商业渠道获得或大肠杆菌重组表达
>40 mg/L |
Butelase 1转肽酶通过植物材料提取
约5 mg/kg |
Omniligase-1可通过商业渠道获得或枯草芽孢杆菌重组表达
>500 mg/L |
—— | |
| 底物活化 | 底物无需活化 | 底物无需活化 |
多肽C端活化为 氧酯或硫酯 |
多肽C端活化为 硫酯 |
|
|
连接 位点 序列 |
C端 | LPXT-G | N/D-HV | X4X3X2X1—,X2外均避免Pro,X4偏好疏水&芳香族氨基酸 | 无限制 |
| N端 | (G)n |
—X1X2,其中X1避免Pro和 酸性氨基酸,X2一般为ILVC |
—X1’X2’均避免Pro | Cys | |
| 连接“疤痕” | LPXT(G)n | 一个Asn/Asp残基 | 无痕(X4X3X2X1 X1’X2’) | 一个Cys残基 | |
|
应用 范围 |
多肽首 尾环化 |
短肽(大于19个氨基酸残基)和蛋白质皆可环化 | 短肽(大于9个氨基酸残基)和蛋白质皆可环化 | 环化多肽一般不超过40个残基 | 环化多肽一般不超过40个残基 |
|
蛋白末 端修饰 |
蛋白质N端与C端皆可 | 蛋白质N端与C端皆可 | 大多用于蛋白质N端修饰鲜有C端修饰 | 蛋白质N端与C端皆可 | |
|
蛋白质 全合成 |
尚无应用报道 | 尚无应用报道 | 可以应用 | 广泛应用 | |
《合成生物学》|从药物多肽到蛋白质全合成:酶促拼接的方法原理与前沿应用
参考文献
参考文献
[1]
LAU J L, DUNN M K.
Therapeutic peptides: historical perspectives, current development trends, and future directions
[J]. Bioorganic & Medicinal Chemistry, 2018, 26(10): 2700-2707.
[本文引用: 1]
[2]
XIAO Y, JIE M, LI B, et al.
Peptide-based treatment: a promising cancer therapy
[J]. Journal of Immunology Research, 2015,2015: 761820.
[3]
VADEVOO S M P, GURUNG S, KHAN F, et al.
Peptide-based targeted therapeutics and apoptosis imaging probes for cancer therapy
[J]. Archives of Pharmacal Research, 2019, 42(2): 150-158.
[本文引用: 1]
[4]
TOPLAK A, NUIJENS T, QUAEDFLIEG P J L M, et al.
Peptiligase, an enzyme for efficient chemoenzymatic peptide synthesis and cyclization in water
[J]. Advanced Synthesis & Catalysis, 2016, 358: 2140-2147.
[本文引用: 2]
[5]
ZORZI A, DEYLE K, HEINIS C.
Cyclic peptide therapeutics: past, present and future
[J]. Current Opinion in Chemical Biology, 2017, 38: 24-29.
[本文引用: 1]
[6]
CHOW H Y, ZHANG Y, MATHESON E, et al.
Ligation technologies for the synthesis of cyclic peptides
[J]. Chemical Reviews, 2019, 119(17): 9971-10001.
[本文引用: 1]
[7]
WEEKS A M, WELLS J A.
Subtiligase-catalyzed peptide ligation
[J]. Chemical Reviews, 2020, 120(6): 3127-3160.
[本文引用: 3]
[8]
HOUEN G. Peptide antibodies[M]. New York: Humana Press, 2015: 33-50.
[本文引用: 1]
[9]
BEHRENDT R, WHITE P, OFFER J.
Advances in Fmoc solid-phase peptide synthesis
[J]. Journal of Peptide Science, 2016, 22(1): 4-27.
[本文引用: 1]
[10]
SCHMIDT M, TOPLAK A, QUAEDFLIEG P J L M, et al.
Enzyme-mediated ligation technologies for peptides and proteins
[J]. Current Opinion in Chemical Biology, 2017, 38: 1-7.
[本文引用: 3]
[11]
ZHANG Y, PARK K, SUAZO K F, et al.
Recent progress in enzymatic protein labelling techniques and their applications
[J]. Chemical Society Reviews, 2018, 47(24): 9106-9136.
[本文引用: 1]
[12]
AGOURIDAS V, MAHDI O E, DIEMER V, et al.
Native chemical ligation and extended methods: mechanisms, catalysis, scope, and limitations
[J]. Chemical Reviews, 2019, 119(12): 7328-7443.
[本文引用: 2]
[13]
CONIBEAR A C, WATSON E E, PAYNE R J, et al.
Native chemical ligation in protein synthesis and semi-synthesis
[J]. Chemical Society Reviews, 2018, 47(24):9046-9068.
[本文引用: 3]
[14]
TAM A, SOELLNER M B, RAINES R T.
Water-soluble phosphinothiols for traceless Staudinger ligation and integration with expressed protein ligation
[J]. Journal of the American Chemical Society, 2007, 129(37): 11421-11430.
[本文引用: 1]
[15]
PUSTERLA I, BODE J W.
The mechanism of the a-ketoacid-hydroxylamine amide-forming ligation
[J]. Angewandte Chemie International Edition, 2012, 51: 513-516.
[本文引用: 1]
[16]
ZHANG Y F, XU C, LAM H Y, et al.
Protein chemical synthesis by serine and threonine ligation
[J]. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(17): 6657-6662.
[本文引用: 1]
[17]
KULKARNI S S, WATSON E E, PREMDJEE B, et al.
Diselenide-selenoester ligation for chemical protein synthesis
[J]. Nature Protocols, 2019, 14: 2229-2257.
[本文引用: 3]
[18]
DAWSON P E, MUIR T W, CLARK-LEWIS I, et al.
Synthesis of proteins by native chemical ligation
[J]. Science, 1994, 266(5186):776-779.
[本文引用: 1]
[19]
ZHENG J, TANG S, QI Y, et al.
Chemical synthesis of proteins using peptide hydrazides as thioester surrogates
[J]. Nature Protocols, 2013, 8: 2483-2495.
[本文引用: 1]
[20]
FLOOD D T, HINTZEN J C J, BIRD M J, et al.
Leveraging the Knorr pyrazole synthesis for the facile generation of thioester surrogates for use in native chemical ligation
[J]. Angewandte Chemie International Edition, 2018, 57: 11634-11639.
[本文引用: 1]
[21]
MUIR T W, SONDHI D, COLE P A.
Expressed protein ligation: a general method for protein engineering
[J]. Proceedings of the National Academy of Sciences of the United States of America, 1998, 95(12): 6705-6710.
[本文引用: 1]
[22]
MALINS L R, PAYNE R J.
Recent extensions to native chemical ligation for the chemical synthesis of peptides and proteins
[J]. Current Opinion in Chemical Biology, 2014, 22: 70-78.
[本文引用: 1]
[23]
VILA-PERELLÓ M, LIU Z, SHAH N H, et al.
Streamlined expressed protein ligation using split inteins
[J]. Journal of the American Chemical Society, 2013, 135: 286-292.
[本文引用: 1]
[24]
MAZMANIAN S K, LIU G, TON-THAT H, et al.
Staphylococcus aureus sortase, an enzyme that anchors surface proteins to the cell wall
[J]. Science, 1999, 285(5428): 760-763.
[本文引用: 1]
[25]
ATONS J M, CHEW G L, GUIMARAES C P, et al.
Site-specific N-and C-terminal labeling of a single polypeptide using sortases of different specificity
[J]. Journal of the American Chemical Society, 2009, 131(31): 10800-10801.
[本文引用: 1]
[26]
NUIJENS T, TOPLAK A, SCHMIDT M, et al.
Natural occurring and engineered enzymes for peptide ligation and cyclization
[J]. Frontiers in Chemistry, 2019, 7: 829.
[本文引用: 8]
[27]
MAO H, HART S A, SCHINK A, et al.
Sortase-mediated protein ligation: a new method for protein engineering
[J]. Journal of the American Chemical Society, 2004, 126(9): 2670-2671.
[本文引用: 1]
[28]
TANAKA T, YAMAMOTO T, TSUKIJI S, et al.
Site-specific protein modification on living cells catalyzed by sortase
[J]. ChemBioChem, 2008, 9: 802-807.
[本文引用: 3]
[29]
YAMAMOTO T, NAGAMUNE T.
Expansion of the sortase-mediated labeling method for site-specific N-terminal labeling of cell surface proteins on living cells
[J]. Chemical Communications, 2009, 9: 1022-1024.
[本文引用: 1]
[30]
HESS G T, CRAGNOLINI J J, POPP M W, et al.
M13 bacteriophage display framework that allows sortase-mediated modification of surface-accessible phage proteins
[J]. Bioconjugate Chemistry, 2012, 23(7): 1478-1487.
[本文引用: 3]
[31]
VAN'T HOF W, MAŇÁSKOVÁ S H, VEERMAN E C I, et al.
Sortase-mediated backbone cyclization of proteins and peptides
[J]. Biological Chemistry, 2015, 396(4): 283-293.
[本文引用: 1]
[32]
BOLSCHER J G M, OUDHOFF M J, NAZMI K, et al.
Sortase A as a tool for high-yield histatin cyclization
[J]. The FASEB Journal, 2011, 25(8): 2650-2658.
[本文引用: 3]
[33]
STANGER K, MAURER T, KALUARACHCHI H, et al.
Backbone cyclization of a recombinant cystine-knot peptide by engineered Sortase A
[J]. FEBS Letters, 2014, 588(23): 4487-4496.
[本文引用: 1]
[34]
ZHANG J, YAMAGUCHI S, NAGAMUNE T.
Sortase A-mediated synthesis of ligand-grafted cyclized peptides for modulating a model protein-protein interaction
[J]. Biotechnology Journal, 2015, 10: 1499-1505.
[本文引用: 1]
[35]
SCHMOHL L, SCHWARZER D.
Sortase-mediated ligations for the site-specific modification of proteins
[J]. Current Opinion in Chemical Biology, 2014, 22: 122-128.
[本文引用: 1]
[36]
SCHMIDT M, TOPLAK A, QUAEDFLIEG P J L M, et al.
Enzyme-catalyzed peptide cyclization
[J]. Drug Discovery Today. Technologies, 2017, 26: 11-16.
[本文引用: 1]
[37]
THOMPSON R E, STEVENS A J, MUIR T W.
Protein engineering through tandem transamidation
[J]. Nature Chemistry, 2019, 11: 737-743.
[本文引用: 1]
[38]
LI Y, LI Y, PAN M, et al.
Irreversible site-specific hydrazinolysis of proteins by use of sortase
[J]. Angewandte Chemie International Edition, 2014, 53: 2198-2202.
[本文引用: 1]
[39]
NGUYEN G K T, WANG S J, QIU Y B, et al.
Butelase 1 is an Asx-specific ligase enabling peptide macrocyclization and synthesis
[J]. Nature Chemical Biology, 2014, 10(9): 732-738.
[本文引用: 1]
[40]
NGUYEN G K T, KAM A, LOO S, et al.
Butelase 1: a versatile ligase for peptide and protein macrocyclization
[J]. Journal of the American Chemical Society, 2015, 137(49): 15398-15401.
[本文引用: 2]
[41]
NGUYEN G K T, QIU Y B, CAO Y, et al.
Butelase-mediated cyclization and ligation of peptides and proteins
[J]. Nature Protocols, 2016, 11(10): 1977-1988.
[本文引用: 1]
[42]
HEMU X, QIU Y B, NGUYEN G K T, et al.
Total synthesis of circular bacteriocins by Butelase 1
[J]. Journal of the American Chemical Society, 2016, 138(22): 6968-6971.
[本文引用: 3]
[43]
HEMU X, ZHANG X H, TAM J P.
Ligase-controlled cyclo-oligomerization of peptides
[J]. Organic Letters, 2019, 21(7): 2029-2032.
[本文引用: 1]
[44]
NGUYEN G K T, CAO Y, WANG W, et al.
Site-specific N-terminal labeling of peptides and proteins using butelase 1 and thiodepsipeptide
[J]. Angewandte Chemie International Edition, 2015, 54: 15694-15698.
[本文引用: 3]
[45]
BI X, YIN J, NGUYEN G K T, et al.
Enzymatic engineering of live bacterial cell surfaces using butelase 1
[J]. Angewandte Chemie International Edition, 2017, 56: 7822-7825.
[本文引用: 3]
[46]
CAO Y, NGUYEN G K T, CHUAH S, et al.
Butelase-mediated ligation as an efficient bioconjugation method for the synthesis of peptide dendrimers
[J]. Bioconjugate Chemistry, 2016, 27(11): 2592-2596.
[本文引用: 1]
[47]
CAO Y, NGUYEN G K T, TAM J P, et al.
Butelase-mediated synthesis of protein thioesters and its application for tandem chemoenzymatic ligation
[J]. Chemical Communications, 2015, 51(97): 17289-17292.
[本文引用: 3]
[48]
NUIJENS T, SCHMIDT M. Butelase 1-mediated ligation of peptides and proteins [M]. New York: Humana Press, 2019: 83-109.
[本文引用: 1]
[49]
HARRIS K S, DUREK T, KAAS Q, et al.
Efficient backbone cyclization of linear peptides by a recombinant asparaginyl endopeptidase
[J]. Nature Communications, 2015, 6: 10199.
[本文引用: 1]
[50]
YANG R, WONG Y H, NGUYEN G K T, et al.
Engineering a catalytically efficient recombinant protein ligase
[J]. Journal of the American Chemical Society, 2017, 139: 5351-5358.
[本文引用: 1]
[51]
POLGARF L, BENDER M L.
The reactivity of thiol-subtilisin, an enzyme containing a synthetic functional group
[J]. Biochemistry, 1967, 6(2): 610-620.
[本文引用: 1]
[52]
NAKATSUKA T, SASAKI T, KAISER E T.
Peptide segment coupling catalyzed by the semisynthetic enzyme thiolsubtilisin
[J]. Journal of the American Chemical Society, 1987, 109(12): 3808-3810.
[本文引用: 1]
[53]
ABRAHMSÉN L, TOM J, BURNIER J, et al.
Engineering subtilisin and its substrates for efficient ligation of peptide bonds in aqueous solution
[J]. Biochemistry, 1991, 30(17): 4151-4159.
[本文引用: 1]
[54]
CHANG T K, JACKSON D Y, BURNIER J P, et al.
Subtiligase: a tool for semisynthesis of proteins
[J]. Proceedings of the National Academy of Sciences of the United States of America, 1994, 91(26): 12544-12548.
[本文引用: 1]
[55]
ATWELL S, WELLS J A.
Selection for improved subtiligases by phage display
[J]. Proceedings of the National Academy of Sciences of the United States of America, 1999, 96(17): 9497-9502.
[本文引用: 1]
[56]
WEEKS A M, WELLS J A.
Engineering peptide ligase specificity by proteomic identification of ligation sites
[J]. Nature Chemical Biology, 2018, 14(1): 50-57.
[本文引用: 1]
[57]
JACKSON D Y, BURNIER J, QUAN C, et al.
A designed peptide ligase for total synthesis of ribonuclease a with unnatural catalytic residues
[J]. Science, 1994, 266(5183): 243-247.
[本文引用: 4]
[58]
JACKSON D Y, BUMIER J P, WELLS J A.
Enzymatic cyclization of linear peptide esters using subtiligase
[J]. Journal of the American Chemical Society, 1995, 117: 819-820.
[本文引用: 1]
[59]
HENAGER S H, CHU N, CHEN Z, et al.
Enzyme-catalyzed expressed protein ligation
[J]. Nature Methods, 2016, 13(11): 925-927.
[本文引用: 1]
[60]
MAHRUS S, TRINIDAD J C, BARKAN D T, et al.
Global sequencing of proteolytic cleavage sites in apoptosis by specific labeling of protein N termini
[J]. Cell, 2008, 134: 866-876.
[本文引用: 4]
[61]
NUIJENS T, TOPLAK A, QUAEDFLIEG P J L M, et al.
Engineering a diverse ligase toolbox for peptide segment condensation
[J]. Advanced Synthesis & Catalysis, 2016, 358(24): 4041-4048.
[本文引用: 2]
[62]
SCHMIDT M, TOPLAK A, QUAEDFLIEG P J L M, et al.
Omniligase-1: a powerful tool for peptide head-to-tail cyclization
[J]. Advanced Synthesis & Catalysis, 2017, 359: 2050-2055.
[本文引用: 1]
[63]
PAWLAS J, NUIJENS T, PERSSON B, et al.
Sustainable, cost-efficient manufacturing of therapeutic peptides using chemo-enzymatic peptide synthesis (CEPS)
[J]. Green Chemistry, 2019, 21: 6451-6467.
[本文引用: 3]
[64]
SCHMIDT M, HUANG Y, OLIVEIRA E F T, et al.
Efficient enzymatic cyclization of disulfide-rich peptides using peptiligases
[J]. ChemBioChem, 2019, 20(12): 1524-1529.
[本文引用: 1]
[65]
RICHELLE G J J, SCHMIDT M, IPPEL H, et al.
A one-pot "triple-c'' multicyclization methodology for the synthesis of highly constrained isomerically pure tetracyclic peptides
[J]. ChemBioChem, 2018, 19(18): 1934-1938.
[本文引用: 1]
[66]
STREEFKERK D E, SCHMIDT M, IPPEL J H, et al.
Synthesis of constrained tetracyclic peptides by consecutive CEPS, CLIPS, and oxime ligation
[J]. Organic Letters, 2019, 21: 2095-2100.
[本文引用: 1]
[67]
SCHMIDT M, TOPLAK A, ROZEBOOM H J, et al.
Design of a substrate-tailored peptiligase variant for the efficient synthesis of thymosin-α1
[J]. Organic & Biomolecular Chemistry, 2018, 16: 609-618.
[本文引用: 1]
[68]
HENNINOT A, COLLINS J C, NUSS J M.
The current state of peptide drug discovery: back to the future?
[J]. Journal of Medicinal Chemistry, 2018, 61: 1382-1414.
[本文引用: 1]
[69]
HUANG P, BOYKEN S E, BAKER D.
The coming of age of de novo protein design
[J]. Nature, 2016, 537: 320-327.
[本文引用: 1]
[70]
MILLS J H, KHARE S D, BOLDUC J M, et al.
Computational design of an unnatural amino acid dependent metalloprotein with atomic level accuracy
[J]. Journal of the American Chemical Society, 2013, 135(36): 13393-13399.
[本文引用: 1]







 南京肽业生物科技有限公司
南京肽业生物科技有限公司
 地址:
地址:
 Email: info@njpeptide.com
Email: info@njpeptide.com
 总机:025-58361106-801
总机:025-58361106-801
